Exploration genomischer Methoden für die Abgrenzung von Arten in Radiationen von Landschnecken
Wir wollen Labormethoden für die Generierung genomischer Daten und analytische Methoden für die Abgrenzung von Arten mit solchen Daten evaluieren und optimieren. Ein besonderer Schwerpunkt in unserem Projekt wird die Evaluierung und Weiterentwicklung von Methoden für die Abgrenzung von nah verwandten Arten sein. Wir werden untersuchen, in wie weit die Artabgrenzung durch einen integrativen Ansatz, der neben genomischen Daten auch morphologische, ökologische und geographische Daten berücksichtigt, verbessert werden kann. Wir werden erkunden, ob die Abgrenzung von nah verwandten Arten durch die Konzentration auf Teilmengen von Loci, die unter Selektion stehen und vermutlich auch Speziationsgene oder Loci, die mit diesen gekoppelt sind, enthalten, verbessert werden kann. Wir werden Artabgrenzungsmethoden, die SNP Daten nutzen, mit Methoden vergleichen, die DNA Sequenzdaten verwenden. Dazu werden wir die Ergebnisse dieser Methoden basierend auf SNP und DNA Sequenzdaten, die mit derselben Labormethode in derselben Artengruppe generiert wurden, vergleichen. Wir wollen die Taxonomie und Evolutionsgeschichte von Radiationen von Schließmundschnecken in frühen Stadien des Artbildungsprozesses durch die Anwendung der erwähnten genomischen Methoden aufklären.
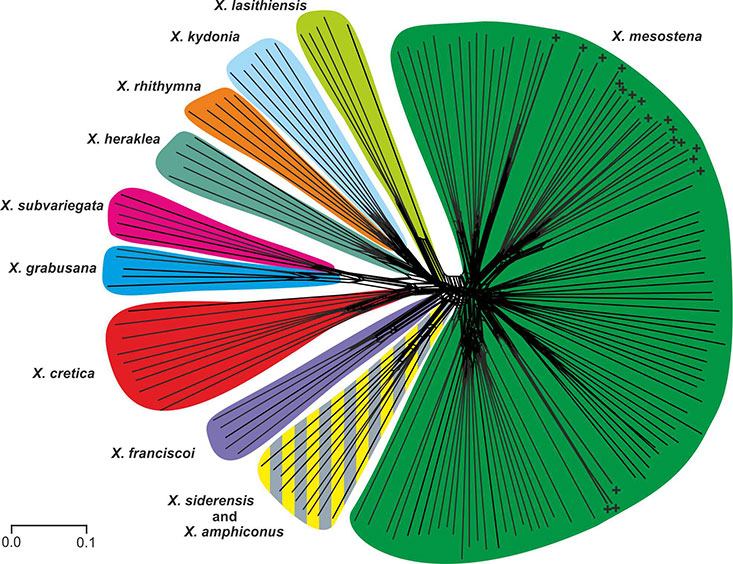
- Doktorarbeit Sonja Bamberger (gefördert im DFG Schwerpunktprogramm 1991 Taxon-OMICS: Neue Herangehensweisen zur Entdeckung und Benennung von Arten und Biodiversität).
- Hausdorf, B. & Hennig, C. 2010. Species delimitation using dominant and codominant multilocus markers. – Syst. Biol., 59 (5): 491–503.
- Sauer, J. & Hausdorf, B. 2012. A comparison of DNA based methods for delimiting species in a Cretan land snail radiation reveals shortcomings of exclusively molecular taxonomy. – Cladistics, 28 (3): 300–316.

